パーキンソン病におけるプロテオミクス解析の有用性は不明だった
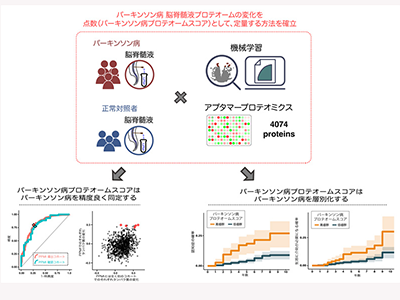
京都大学は8月18日、パーキンソン病に特異的な変化のパターン(パーキンソン病シグネチャー)を同定し、そのパターンを点数(パーキンソン病プロテオミクススコア)として定量化することに成功したと発表した。この研究は、同大大学院医学研究科の月田和人特任助教(帝京大学先端統合研究機構特任研究員、関西電力医学研究所睡眠医学研究部特任研究員兼務)、高橋良輔同教授らの研究グループによるもの。研究成果は、「Neurology」にオンライン掲載されている。
画像はリリースより
(詳細は▼関連リンクからご確認ください)
プロテオミクス解析の進歩により、さまざまな手法が開発されている。その1つに、アプタマープロテオミクスがある。アプタマープロテオミクスでは、数千のタンパク質を、迅速に多サンプルで測定できる。そのため、プロテオミクス解析を大規模サンプルに応用することが可能になってきた。近年、アプタマープロテオミクスを用いて、血漿サンプルから、現在の加齢に関する情報や心血管機能に関する情報のみならず、将来の糖尿病発症リスクや心筋梗塞などの心血管イベントの発症リスクなど、さまざまな情報が抽出できることが示されてきている。一方、パーキンソン病に関しては、プロテオミクス解析をどのように応用すれば良いのか、プロテオミクス解析でどのような有用な情報が得られるのか、不明だった。
1,149人の脳脊髄液をプロテオミクス解析、結果データに機械学習を応用
今回の研究では、国際多施設共同観察研究PPMI(Parkinson’s Progression Markers Initiative)研究のデータを用いた。PPMI研究では、パーキンソン病患者、正常対照者、また、パーキンソン病関連遺伝子変異をもった非パーキンソン病患者などを研究に組み入れ、多くの臨床項目を長期的かつ包括的に評価している。特記すべき点として、脳脊髄液などの生体資料の保存も行っている。近年、1,149人の保存された脳脊髄液に対して、アプタマープロテオミクスが行われ、4,071個のタンパク質が定量された。今回の研究では、このデータに機械学習を応用した。
関連遺伝子変異を「持たない」患者と非患者を精度良く区別
まず、パーキンソン病関連遺伝子変異を持たないパーキンソン病患者279人と正常対照者141人のデータから、パーキンソン病シグネチャーを同定。そのパターンをパーキンソン病プロテオミクススコアとして定量化することに成功した。パーキンソン病プロテオミクススコアは、PPMI研究内部の独立したパーキンソン病関連遺伝子変異を持たないパーキンソン病患者71人と正常対照者35人のデータを精度良く区別することが確認された。全く別のコホート(LRRK2 Cohort Consortium)のパーキンソン病関連遺伝子変異を持たないパーキンソン病患者31人と正常対照者34人のデータも、精度良く区別したという。続いて、PPMI研究内部のパーキンソン病関連遺伝子変異を持つパーキンソン病患者258人とパーキンソン病関連遺伝子変異を持つ非パーキンソン病患者365人のデータも精度良く区別することも検証された。
認知機能低下・運動機能低下の進行も強力に層別化
最後に、パーキンソン病プロテオミクススコアと予後との関連を検討。その結果、パーキンソン病プロテオミクススコアは、認知機能低下の進行、運動機能低下の進行、ともに、強力に層別化したとしている。
今回の研究により、パーキンソン病患者の脳脊髄液プロテオームの特徴を捉える方法論が見出された。また、パーキンソン病では、脳脊髄液プロテオームの変化が疾患早期から生じており、それがその後の病態の進行に深く関わる可能性が高いことが示された。この結果は、プロテオミクス解析を有効に用いることで、早期から患者の層別化が可能であることを示すのみならず、パーキンソン病の脳脊髄液プロテオームの変化を正すことで疾患修飾ができる可能性を示唆する、と研究グループは述べている。
▼関連リンク
・京都大学 プレスリリース



