複数のHLA遺伝子の臨床応用可能なゲノム編集、ヒトiPS細胞ではまだ達成されていない
京都大学iPS細胞研究財団(CiRA_F)は6月13日、HLAホモ接合体iPS細胞において、3つの遺伝子座(HLA-A、HLA-B、CIITA遺伝子)を同時編集するGMP対応のCRISPR-Cas9ゲノム編集のプロトコールを開発したと発表した。この研究は、京都大学iPS細胞研究財団を中心とした研究グループによるもの。研究成果は、「Molecular Therapy – Methods & Clinical Development」にオンライン掲載されている。
画像はリリースより
(詳細は▼関連リンクからご確認ください)
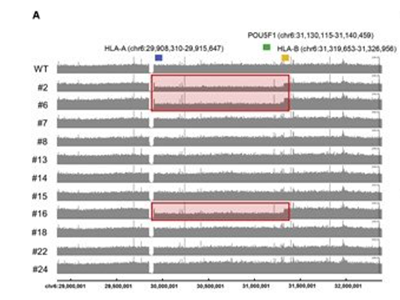
近年、CRISPR-Cas9ゲノム編集技術を用いて、HLA-Cを保持したまま、HLA-A、HLA-BおよびCIITA遺伝子を選択的にノックアウトし、免疫拒絶反応を抑制する新しい戦略が開発された。このようなHLAゲノム編集iPS細胞は、HLA-A、HLA-B、およびHLAクラスⅡが欠失していることで、キラーT細胞およびNK細胞の両方の攻撃を回避できることが示されている。複数の遺伝子をノックアウトするために段階的なゲノム編集を実施した場合、培養期間は長期間に及ぶ。一方、同時に複数の遺伝子を同時にノックアウトする多重ゲノム編集では培養期間を大幅に削減出来るため、細胞培養に伴うゲノム変異等のリスクを最小限に抑えられる点で有利だが、染色体間の大規模なゲノム構造異常の可能性がある。
さらに、HLA遺伝子のゲノム編集では、通常考慮すべきオフターゲット変異リスクに加えて、HLA遺伝子のDNA配列が互いに類似していることから、他の遺伝子座を標的とする場合よりもさらに変異誘発や染色体再配列のリスクが高くなる可能性がある。このため、品質評価方法については、これまで以上に留意する必要があるが、臨床用製造や品質評価方法におけるリスク評価は十分に解析されていなかった。
3遺伝子同時編集で製造過程短縮、遺伝子異常が多く生じるが適切な品質試験で細胞選択可能
今回の研究では、HLA-A、HLA-B、CIITAの3つの遺伝子を同時にゲノム編集したiPS細胞を製造する手法を確立した。Cas9タンパク質と化学合成した2種のgRNA(HLA-AとHLA-Bは共通のgRNAで編集できるよう設計したため1種のgRNAで2つの遺伝子の編集が可能)の複合体をHLAホモ接合体iPS細胞(Ff-I14s04株)にエレクトロポレーションで導入。その後、シングルセルクローニングを行い、ゲノム解析と分化能評価を含む細胞機能アッセイを実施した。
その結果、HLA遺伝子座のゲノム編集は、WGS、核型解析、オプティカルゲノムマッピング分析によって評価した複数のクローンにおいて、複数のゲノム編集標的部位が関わったCNV、染色体転座、複雑なゲノム構造変異を引き起こしていたことが判明した。しかし、研究グループが実施した多角的な品質試験で、これらの変異を十分に追跡・解析することができ、目的としたゲノム編集以外の変異リスクを抑えた細胞を選択することが可能となった。
臨床応用に向けて、ゲノム解析を含む多角的な品質評価試験が重要
今回の研究により、臨床用iPS細胞におけるCRISPR-Cas9を用いた同時多重HLAゲノム編集の課題と解決策について、基盤技術となる成果を得ることができた。これは、ゲノム編集した低免疫原性iPS細胞を臨床応用につなげるために、ゲノム解析を含む、多角的な品質評価試験が重要であることを示しているという。
今回の研究成果に基づいて、臨床用のHLAゲノム編集iPS細胞を早期に提供できるよう、京都大学iPS細胞研究財団(CiRA_F)細胞調製施設で製造を進めていく、としている。
▼関連リンク
・京都大学iPS細胞研究財団 プレスリリース




