ゲノムDNAのヒドロキシメチル化レベル、従来の測定法では煩雑な操作が必要だった
東京工科大学は6月6日、がんや中枢神経疾患のバイオマーカーとして期待される、ゲノムDNAのヒドロキシメチル化レベルを、試薬を混合するだけで簡便かつ迅速に測定できる方法を開発したと報告した。この研究は、同大応用生物学部の吉田亘准教授と松井徹教授らの研究グループによるもの。研究成果は、「Analytical Chemistry」にオンライン掲載されている。
画像はリリースより
(詳細は▼関連リンクからご確認ください)
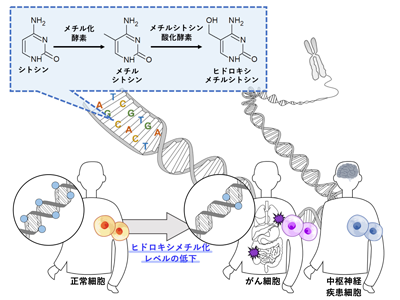
ヒトゲノムDNAのCpG配列中のシトシンは、DNAメチル化酵素によりメチル化され、これにより遺伝子の発現が制御される。DNA脱メチル化反応に関連する酵素としてメチルシトシン酸化酵素が同定されており、この酵素によってメチルシトシンはヒドロキシメチルシトシンへと酸化される。正常細胞では正常なメチル化パターンが形成されているが、がん細胞などの異常な細胞ではゲノムDNA全体のメチル化やヒドロキシメチル化レベルが低下することがわかっている。
DNA修飾状態の異常は、アルツハイマー病などの中枢神経疾患でも報告されていることから、これらの疾病の診断のためのバイオマーカーとしての応用が期待されるが、既存の高速液体クロマトグラフィー法などのメチル化やヒドロキシメチル化レベル測定法では、煩雑な操作が必要となっていた。同研究グループは、これまでに試薬を混合するだけで40分以内にメチル化レベルを測定できる方法を開発しており、本研究では同一のプラットフォームにより、ヒドロキシメチル化レベルを測定する方法を開発することを目的とした。
試薬を混合し、蛍光強度を測定するだけで、40分以内に測定可能
研究グループは、ヒドロキシメチル化レベルを測定するために、ヒドロキシメチル化CpG結合タンパク質であるUHRF2(Ubiquitin like with PHD and ring finger domains 2)のヒドロキシメチル化CpGに結合するタンパク質ドメイン(SRA domain)に着目した。まず、UHRF2 SRAにホタル由来の発光タンパク質であるルシフェラーゼを融合させたタンパク質(UHRF2 SRA-Fluc)を組換え生産した。
また、UHRF2 SRAのメチル化CpGへの結合を阻害するため、メチル化CpG結合タンパク質であるMBD(Methyl-CpG-binding domain)も組換え生産した。次に、DNAインターカレーターとMBDを結合させたヒトゲノムDNAを調製し、UHRF2 SRA-Flucとルシフェラーゼ発光基質を加えた。その結果、UHRF2 SRA-FlucがヒトゲノムDNA中のヒドロキシメチル化CpG部位で発光し、近傍のインターカレーターを励起することが示された。この蛍光強度は、ヒトゲノムDNAのヒドロキシメチル化レベルに依存することも示された。これらの結果から、標的ゲノムDNAにUHRF2 SRA-Fluc、MBD、インターカレーター及びルシフェラーゼ発光基質を混合し、蛍光強度を測定するだけで、ヒドロキシメチル化レベルを40分以内に測定できることが確認された。
この研究により、ゲノムDNAのヒドロキシメチル化レベルを、試薬を混合するだけで簡便かつ迅速に測定できることが示された。「脳におけるヒドロキシメチル化レベルの低下は記憶力の低下に関与することも報告されており、生活習慣病やストレス、老化などによる神経細胞への損傷度やそれに伴う記憶障害の制御機構の解明への貢献が期待される」と研究グループは述べている。
▼関連リンク
・東京工科大学 プレスリリース




