ASDリスク増加を十分に説明する遺伝的変異は特定されていない
東北大学は8月27日、表現型の変数を適切に組み合わせて自閉スペクトラム症(ASD)患者をグループ化(クラスタリング)すると、遺伝的感受性因子を特定するチャンスが増える可能性があることに着目し、人工知能の一つである機械学習の手法を活用して、ASDが異種の疾患の集合体である可能性があることを世界で初めて発見したと発表した。この研究は、同大大学院医学系研究科の栗山進一教授を中心とした研究グループによるもの。研究成果は、「Translational Psychiatry」にオンライン掲載されている。
画像はリリースより
これまで、ある疾患名でまとめられ潜在的に異質な集団を一塊として扱った「患者群全員」と対照群とのゲノムワイド関連解析(GWAS)は多くの疾患で実施されてきたが、ASDについては有意なSNPはみられるものの、その効果は小さく、遺伝的感受性因子を特定したとはいえない状況であると指摘されてきた。しかし、遺伝的要因に関しては、一卵性双生児のASDの一致率は92%、二卵性双生児は10%であることや、きょうだい間のASDの再発リスク比は22と高いことなどから、リスクの増加には遺伝的要因が強く寄与していることが示唆されている。
近年ではデータセットに内在するパターンを明らかにできる人工知能技術である機械学習手法が開発されている。研究グループは、機械学習のクラスタリングアルゴリズムとそれに続くGWASは、新規でより遺伝的に均質な集団、クラスターを明らかにするのではないかという仮説を立てた。
クラスター分析とGWASを組み合わせ、65個の有意な遺伝子座を特定
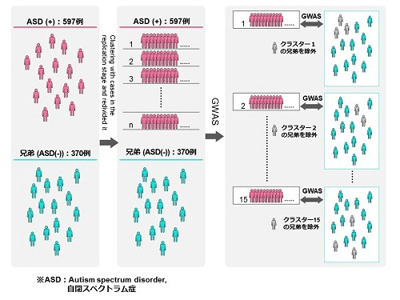
米国のサイモンズ財団(Simons Simplex Collection)からのデータの分譲を受け、クラスター数15のk-meansアゴリズムを使用し、ASDが示す多様な症状とビタミンB6処方歴を変数としてクラスター分析を実施。予備研究として、597例のASD症例と370例の対照のデータセットを使用して、従来のGWASを実施した。2番目のステップでは、クラスタリングの結果に基づいてケースを分割し、各サブグループとコントロール(クラスターベースGWAS)でGWASを行った。
従来のASDという疾患名でまとめられた潜在的に異質な集団を一塊として扱った「患者群全員」と対照群を用いたGWAS手法で行われた予備解析では、有意な関連は観察されなかった。一方、2番目のクラスターに基づいたGWAS(クラスターベースGWAS)では、65個の有意な遺伝子座を特定した。これらの65の遺伝子座のうち、SRRM4遺伝子内に位置するrs11064685は、再現性を検証する解析でも症例と対照で有意に異なる分布を示し、ASDの原因のひとつである可能性が示された。
今回の分析結果は、クラスタリングが比較的均一な病因をもつサブグループを首尾よく特定できる可能性を示唆するもの。研究グループは、「本研究は、データセットが複数の異種のサブグループで構成されている場合、均質な個体の数がはるかに少ないサブグループでさえ、影響が大きく統計学的に有意な遺伝的要因を検出できることを示した」と、述べている。
▼関連リンク
・東北大学 プレスリリース




