ウイルスゲノムの安定化構造を特定すれば、治療標的分子探索につながる
東京大学アイソトープ総合センターは5月12日、多数種類のウイルス由来RNA配列の安定化活性を迅速かつ網羅的に調べる新技術を開発したと発表した。この研究は、同センターの脇田寛泰大学院生(研究当時)、川田健太郎特任助教、山地雄大大学院生、和田洋一郎教授、秋光信佳教授、筑波大学大学院人間総合科学研究科の服部恵美大学院生、同大医学医療系・人工知能科学センターの土屋貴穂助教、尾崎遼准教授らの研究グループによるもの。研究成果は、「Biochemical and Biophysical Research Communications」に掲載されている。
画像はリリースより
ウイルスは、遺伝情報物質であるRNAやDNAなどの核酸分子と、これらを取り巻くタンパク質の殻から構成される。細胞に感染したウイルスは、自らのRNAやDNAを細胞内に送り込み、宿主細胞の複製機構やタンパク質合成機構を利用することで増殖する。宿主細胞は、これらのウイルスの増殖を抑制するために、入り込んだRNAやDNAを分解する仕組み(核酸分解機構)を持っており、この仕組みが自然免疫応答の一部を担っている。一方で、ウイルスは、さまざまな分解抑制機構を介して自らのRNAやDNAを安定化することで、宿主細胞が持つ核酸分解機構を逃れる。ウイルスが持つこれらの核酸安定化機構は進化的に保存されており、その感染力や病原性を決める重要な因子となっている可能性が指摘されてきた。したがって、ウイルスのRNAやDNA分子の安定化を引き起こす配列や高次構造、あるいは、これらの配列に結合するタンパク質を明らかにすることで、ウイルス感染症をより効率的に治療するための標的分子を探索することが可能となる。しかしながら、どのようなウイルス配列が細胞の中でのウイルスRNAやDNA分子の安定化を引き起こすかを、迅速かつ大規模に調べる方法はなかった。
新技術「Fate-seq」を開発し、SARSコロナで進化的保存性の高い2種類の配列を同定
研究グループは、この問題に対処すべく、多数種類のウイルス由来RNA配列の安定化活性を網羅的に調べる新技術「Fate-seq」を開発した。Fate-seqでは、遺伝物質であるRNAもしくはDNAを断片化し、これらの断片を組み込んだRNA分子を人工的に合成する。さらに合成したRNA分子を生細胞に取り込ませ、一定時間経過後の残存数を次世代シーケンサにより定量することで、どのようなウイルス配列が安定化活性を示すのかを網羅的に明らかにすることが可能となる。今回、研究グループは、重症急性呼吸器症候群(SARS)の原因となるSARSコロナウイルス(SARS-CoV)を含む26種類のウイルスからウイルス配列を生成し、安定化活性を持つ計625種類の配列を同定した。
同定された安定化配列を調査したところ、SARSコロナウイルスに由来する21種類の安定化配列が含まれていた。これは、今回検討したRNAウイルスの中で最多。そこで、これらのSARSコロナウイルスに由来する安定化配列が、コロナウイルス科のさまざまなウイルスでどの程度保存されているのかを確認したところ、特に進化的保存性の高い2種類の配列が同定された。
保存配列は新型コロナでより強固な可能性、核酸医薬開発の基盤整備へ
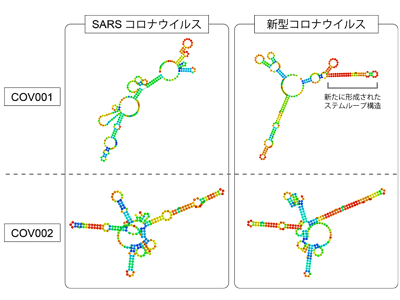
これらの安定化配列は、新型コロナウイルス感染症(COVID-19)の原因となる新型コロナウイルス(SARS-CoV-2)との間でも、92.7%および84.6%と高い塩基保存性を示すことが確認された。さらに、これらのRNA配列の形(RNA二次構造)を予測したところ、片方の配列(COV001)では、新型コロナウイルスにおいて2つの塩基が変異を起こした結果、RNA安定化活性を高めるステムループ構造が新たに形成される可能性が示唆された。また、もう一方の配列(COV002)では、SARSコロナウイルスと新型コロナウイルスの両方で、ステムループ構造が多数形成されることが予測された。これらの結果は、同定された安定化配列が、SARSコロナウイルスと新型コロナウイルスの両方においてRNA分子の安定性を高める働きをすると同時に、新型コロナウイルスではより強固な安定化配列が形成されている可能性を示唆している。
細胞内に入り込んだウイルスをどのように制御するのかは、ウイルス感染症に対する治療法を考えるうえで重要な問題となる。開発したFate-seqは、ウイルスに由来するRNA配列のなかで、RNA安定化活性に強く影響する核酸配列を、迅速かつ網羅的に決定するための強力な戦略となり得る。RNA安定化部位においては、RNA結合タンパク質と呼ばれる一群のタンパク質が結合し、その安定性を高めることが知られている。今回の研究で同定されたRNA安定化部位に結合するRNA結合タンパク質を同定することで、細胞内でのウイルスの安定性を決定する機構を明らかにすることが可能となる。これらのRNA結合タンパク質の活性を人工的に制御することで、より効果的なウイルス治療薬の設計が期待される。さらに、近年DNAやRNAを基本骨格とし、通常の低分子化合物では標的にすることが困難な細胞内RNAに直接作用する核酸医薬が注目されている。Fate-seqにより同定した安定化配列を核酸医薬に組み込むことで、持続時間の制御に利用することも可能だ。研究グループは今後、ウイルスRNA配列情報を利用した核酸医薬の開発基盤を整備する計画だとしている。
▼関連リンク
・東京大学アイソトープ総合センター トピックス



