従来のウイルス感染病態把握は、複数の解析法を組み合わせて実施
熊本大学は10月15日、ヒトT細胞白血病ウイルス(HTLV-1)に感染した細胞DNAに存在するウイルス情報を網羅的かつ高精度に取得する、新しい解析方法を確立したと発表した。この研究は、ヒトレトロウイルス学共同研究センター熊本大学キャンパスの佐藤賢文教授と、佐賀大学の勝屋弘雄助教、鹿児島大学の宇都宮與客員教授、聖マリアンナ医科大学の山野嘉久教授らの共同研究グループによるもの。研究成果は、「Cell Reports」に掲載されている。
画像はリリースより
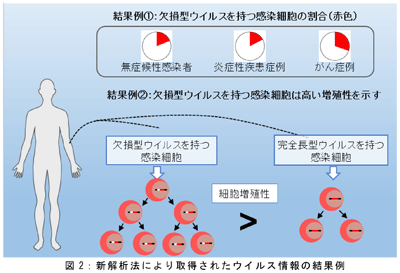
日本に約80万人の感染者が存在するとされるHTLV-1は、感染者の一部にがんや慢性炎症性疾患を引き起こすことが知られている。HTLV-1にはウイルスが感染したヒト細胞のDNAにウイルスDNAを組み込ませる特性があり、それがウイルス病原性の起点となっている。従来、HTLV-1感染者におけるウイルス感染病態を高い精度で把握するためには、ウイルスDNAの全長塩基配列(欠損型ウイルスの有無)、ウイルスDNAの構造、ウイルスDNAの組み込み部位、感染した細胞の増殖度などの情報を取得するために、複数の解析法を組み合わせて行う必要があった。
新手法開発し、日本人感染者約100人の網羅的ウイルス情報を取得
今回研究グループは、ウイルスに対するDNAプローブと次世代シークエンサーを活用した新手法を開発。この手法で、それらのウイルス情報を網羅的かつ高精度に解析することが可能になることを見出した。
さらに、日本人の感染者約100人の網羅的かつ詳細なウイルス情報の取得を実施。これによって得られたウイルス情報は、論文としてオープンアクセスジャーナルで広く公開されるとともに、公的なデータベースに登録しその情報が広く発信され、今後のHTLV-1研究推進や関連疾患の診断向上に資することが期待される。研究グループは、「今回の研究手法はHTLV-1感染症に限らず、他のがんに関連するウイルス感染症などにも応用できる可能性があり、今後のさらなる展開が期待される」と、述べている。
▼関連リンク
・熊本大学 プレスリリース






