ゲノムなど非画像データを深層学習で扱う方法を考案
理化学研究所(理研)は8月6日、人工知能技術の一つである「深層学習」で扱えるように、ゲノミクスデータなどの非画像データを画像データに変換する方法を開発したと発表した。この研究は、同生命医科学研究センター医科学数理研究チームの角田達彦チームリーダー(東京大学大学院理学系研究科生物科学専攻医科学数理研究室教授、東京医科歯科大学難治疾患研究所医科学数理分野教授)らの国際共同研究グループによるもの。研究成果は「Scientific Reports」に8月6日付で掲載された。
画像はリリースより
ゲノミクスデータは、病気などの個人差の解析・診断に役立つが、数万から数千万の変数を持つ超高次元データであることが多く、伝統的な統計学では解析が難しい課題が多く存在する。これを克服するために、近年では「機械学習」に期待が集まっている。研究グループは、特徴抽出を自動的に行えないかと考え、機械学習の中でも、現在の人工知能技術の主流の一つであり、特徴抽出能力を自前で持つ「深層学習」に着目した。深層学習を行うモデルの一つである「畳み込みニューラルネットワーク(CNN)」では、サンプルは画像の形で入力し、隠れ層によって特徴抽出と分類を行う。データから自動的に特徴を導き出すため、追加の特徴抽出手法は必要ない。
ゲノミクスデータなどの多くのデータは非画像形式であり、隣り合う変数同士では明確な関係性が見られない場合が多くある。CNNは入力として画像データを必要とするため、ゲノミクスデータなどを直接は使用できない。ただし、データ同士の関連を適切に考慮しつつ非画像データを画像データに変換できれば、CNNを使って特徴抽出と学習ができ、典型的な機械学習よりも分類性能を向上させることが期待される。そのために、研究グループは今回、画素としての変数の配置をうまく行う方法を考えた。
開発した「ディープインサイト法」、従来法より高精度
研究グループは、データを使って診断や予測のためのクラス同定・分類を高精度に行うためには、「適切な変数(画素)の配置」、「特徴抽出」、そして「適切な分類モデルの構築」、という3つのステップを経る必要があると考え、それらを全て統合した「ディープインサイト法」を提案した。変数の配置に関して、近傍情報を無視して独立に変数を扱うよりも、類似した変数や生データをクラスタとして変換する方が、周囲の重要な情報を互いに補完・統合できるため、信頼性が高くなる。そこで、ディープインサイト法では最初に、似た変数をまとめて配置し、異なるものを離して配置することによって、隣接する変数をまとめて画素集合のように使えるようにした画像を作成。具体的には、学習セットにt-SNEやカーネル主成分分析(kPCA)などの類似度計算手法や次元圧縮手法を用いて、2次元平面を得ることで、各変数の位置(点)を決める。CNNに入力するために、全ての点を含む最小の長方形を見つけ、回転させて水平・垂直の形式にし、各座標をピクセルにマッピングする。変数ベクトルを画像に変換すると、CNNで特徴抽出をし、分類や予測することが可能になる。
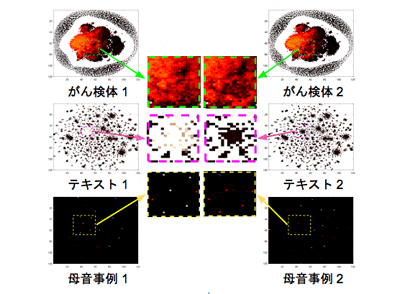
研究グループは次に、ディープインサイト法の検証を試みるため、「遺伝子発現データ」、「テキストデータ」、「母音データ」、「2つの人工データ」のセットを用いて、既存の最先端の分類手法であるランダムフォレスト法、決定木、アダブースト法などと結果を比較した。特に遺伝子発現データは、TCGAからの公共データセットであり、10種類のがんに対応するRNA-seq遺伝子発現データの6,216のサンプルで、各サンプルには6万483の遺伝子発現値(変数)が付与されている。
検証の結果、RNA-seqデータのテストセットを用いたとき、ランダムフォレスト法では分類精度96%だったのに対し、ディープインサイト法では99%を達成した。母音データでは、ランダムフォレスト法の分類精度90%に対してディープインサイト法は97%、テキストデータでは、ランダムフォレスト法の分類精度90%に対し、ディープインサイト法は92%の精度を達成した。残りの2つの人工データセットでも同様の結果だった。さらに、5つのデータセット全てを用いて平均分類精度を計算した結果、既存の技術のうち最高だったランダムフォレスト法の86%に対し、ディープインサイト法は95%の平均分類精度を記録し、はるかに優れたものであることがわかった。
今回開発された手法により、非画像データの解析に対しても、深層学習、特にCNNの能力を活用することができる。また、DNA配列やタンパク質配列、RNA-Seqなどのさまざまなオミクスデータを深層学習によって解析できる可能性がある。現時点で同手法は、入力が単層で2次元行列型のCNNを想定しているが、これを複数の層を組み込むように拡張すれば、例えば、遺伝子発現、メチル化、突然変異などのマルチオミクスデータを扱う問題にも適用することが可能。「深層学習がより利活用されることにより、医学・生命科学の複雑なデータが紐解かれ、将来の個人ごとの診断や予測に役立てられることが期待できる」と、研究グループは述べている。
▼関連リンク
・理化学研究所 プレスリリース



