すべての薬物やヒト細胞の組み合わせに対する網羅的な観測を目指した研究
九州工業大学は7月8日、多様なヒト細胞における薬物応答の遺伝子発現パターンを高精度に予測する新たな情報技術を開発したと発表した。この研究は、同大大学院情報工学研究院の山西芳裕教授らの研究グループが、理化学研究所革新知能統合研究センターのZHAO Qibinユニットリーダーらの研究グループ、田部井靖生ユニットリーダーと共同で行ったもの。研究成果は、英国科学誌「Bioinformatics」のオンライン版に同日付で公開された。
画像はリリースより
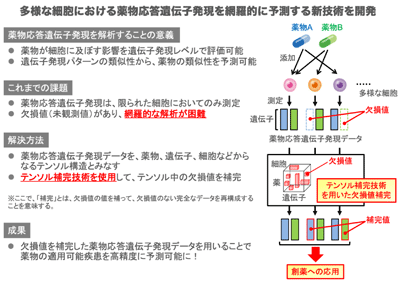
ヒト細胞における薬物応答の遺伝子レベルでの解明は創薬の重要課題である。近年、さまざまなヒト細胞における薬物応答遺伝子発現データが測定されるようになってきた。しかし、薬物応答遺伝子発現データの取得には、多大のコストと時間を要するため、すべての薬物やヒト細胞の組み合わせに対して網羅的に観測することは困難だ。そのため、薬物応答遺伝子発現データは多くの欠損値や未観測値を含んでおり、創薬現場では網羅的な解析を行う上で障害となっていた。
薬物応答を示す遺伝子発現パターンを高精度で予測する機械学習手法を開発
今回の研究では、薬物応答遺伝子発現データを、薬物、遺伝子、細胞、時系列からなるテンソル構造と捉え、新規のテンソル分解手法である「Tensor-train weighted optimizationアルゴリズム」を用いて、さまざまな細胞における未観測の薬物応答を予測する機械学習手法を開発。これを薬物の効能予測に応用し、開発手法が効能予測の性能の向上に大きく寄与することを示した。
まず、薬物、細胞、遺伝子からなる3階のテンソル構造である薬物応答遺伝子発現データにおける未観測値予測性能を評価するため、観測値を人工的に欠損値と見なし、観測値を正しく再現できるか調査。その結果、提案手法は、従来手法に比べて最大約1.5倍の精度で欠損値を補完することができることを見出した。また、薬物応答遺伝子発現パターンは時間的にも変化するので、薬物、細胞、遺伝子、時系列からなる4階のテンソルを構築し、同様の性能評価を実施。その結果、提案手法は、特に高階テンソル構造のデータに対する有効性を示した(従来手法に比べて最大約2倍の精度向上)。
次に、欠損値を補完した薬物応答遺伝子発現データを、薬物の効能の予測に応用。その結果、欠損値が多い細胞ほど、効能予測の性能が大きく向上し、最大約20%性能が向上することを示した。さらに、1,483個の薬物を16種類のヒト細胞に添加して得られた薬物応答遺伝子発現データに提案手法を適用し、駆虫薬であるニクロサミドの成人T細胞白血病に対する効能など、さまざまな疾患に効能があると期待される薬物を選び出した。その新しく予測された薬物の効能についての妥当性は、近年の文献や臨床報告で確認できた。
今後は、アルゴリズムを改善して予測の信頼性や計算効率を向上させていくとともに、既承認薬だけでなく合成化合物や天然化合物などあらゆる化合物にも解析対象を広げ、さまざまな疾患に対する医薬品候補化合物を探索していく予定だという。また、特定の遺伝子を過剰発現またはノックダウンしたときの細胞状態のデータや疾患患者のマルチオミクスデータなど、テンソル構造として解釈することができるさまざまな医薬ビッグデータにも開発手法を適用し、創薬応用や医療応用につなげていく予定としている。
▼関連リンク
・九州工業大学 ニュースリリース




