より良い創薬標的の選定にCRISPRスクリーニングを応用
京都大学は5月7日、ゲノム編集技術CRISPR-Cas9を用いた遺伝子スクリーニング法を30がん種にわたる300以上のがん細胞株に適用し、新しい治療薬のターゲットとなる可能性のある約600遺伝子を発見したと発表した。この研究は、同大ウイルス・再生医科学研究所の遊佐宏介教授らの研究グループによるもの。研究成果は、英国の科学雑誌「Nature」にオンライン掲載されている。
がん分子標的薬の開発には多額の費用と長い時間を必要とし、また9割の新薬開発計画は途中で中止となる。このような厳しい状況である理由のひとつに、期待された効果が臨床試験で得られないということがある。したがって、新薬開発の成功率を上げ、がん患者により多くの新薬を届けるためにも、最初により良い創薬標的を選定することが重要といえる。
研究グループは、2014年にゲノム編集技術CRISPR-Cas9を用いた全遺伝子スクリーニング法(CRISPRスクリーニング法)を開発し、発表した。これはヒトが持つタンパク質をコードする全2万遺伝子を個別に破壊し、その変異細胞が示す異常を調べる手法。がん細胞株に適用することで、どの遺伝子ががん細胞の増殖に必要であるのかを調べることができる。そして、増殖に必要な遺伝子の働きを止める薬を作れば、がんの増殖を止めることができ、がんの治療薬となると考えられる。
画像はリリースより
優先スコアを付けて各遺伝子の創薬可能性を比較
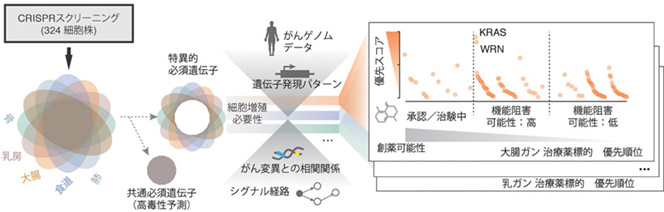
今回、30がん種にわたる324細胞株においてCRISPRスクリーニングを実施。各がん細胞株における細胞の生存・増殖に必須の遺伝子のリストを得て、約7,000個の遺伝子が少なくとも1つの細胞株の増殖に必要であることが明らかとなった。これらの遺伝子は全てその機能を抑制するとがん細胞の増殖を止められる可能性があるが、この膨大な候補の中から、毒性は低く、がん抑制効果は高く、また治療の対象となるがん種がはっきりしている創薬の候補遺伝子を絞り込まなくてはいけない。
そこで研究グループは、得られたデータと過去に蓄積されたがんゲノムデータを使ってさまざまなコンピューター解析を行い、解析結果を統合することで、より有用性の高いがん創薬ターゲットに優先順位をつけるシステムを構築。まず、がん種ごとに解析が行われ、各遺伝子に優先スコアがつけられる。このステップでは、共通必須遺伝子(細胞毒性が高いと予測され創薬対象から外した方が良い)の同定と、がん変異遺伝子と細胞増殖に与える影響の相関関係(バイオマーカーを持つ患者に効果の高い薬の指標となる)が特に重要となる。こうして付けられた優先スコアと各遺伝子の創薬可能性を比較することで、どの遺伝子に対して創薬を行うのが適切であるかの情報を得ることができる。
マイクロサテライト不安定性がんの治療薬候補を同定
研究グループは、今回得られた創薬優先順位の高い遺伝子の中から、Werner症候群RecQヘリケース (WRN) を見出した。この遺伝子はマイクロサテライト不安定性を示すがん(大腸がん、胃がん、卵巣がん等)においてのみ、がん細胞の増殖に必要であることがわかった。マイクロサテライト不安定性は、DNA損傷修復経路の一つ、ミスマッチ修復に関わる遺伝子の変異が原因となっている。
解析を進めた結果、この依存性は WRNタンパク質が持つヘリケース活性によるものであることが明らかとなり、また、異種移植モデルにおいてWRN遺伝子を破壊すると腫瘍の退縮が見られることが実験的に確認され、WRNのヘリケース活性を阻害するような薬がマイクロサテライト不安定性を示すがんに対する分子標的薬となる可能性が示された。
今回取得された、各がん細胞株の必須遺伝子プロファイル、優先順位スコア等は全て専用のウェブサイトで公開されている。今回発見されたその他の候補遺伝子を詳しく解析することで、さらに有望な創薬ターゲット候補が見つかり、より多くの治療選択が可能となることが期待されると、研究グループは述べている。
▼関連リンク
・京都大学 研究成果



