レプトスピラの分布や生息については多くが未解明
琉球大学は4月25日、保菌動物の尿から川や土壌を経由してヒトに感染する病原体レプトスピラについて、河川の水から直接DNAを検出することに成功、さらに、レプトスピラと同時に出現する動物のDNAを分析することによって、レプトスピラの保菌動物の候補(イノシシやオオコウモリなど)を推定できることを示したと発表した。この研究は、琉球大学・戦略的研究プロジェクトセンターの佐藤行人特命講師、同大学院・医学研究科のトーマ・クラウディア准教授、新潟大学・医学部保健学科のサトウ恵助教、神戸大学大学院・人間発達環境学研究科の源利文准教授らの研究グループによるもの。研究成果は、英国の学術雑誌「Scientific Reports」に掲載されている。
画像はリリースより
レプトスピラは、哺乳類を中心に多くの動物に感染する人獣共通感染症の病原細菌。保菌動物の尿から河川や土壌を経由してヒトに感染し、発熱や、時には重篤な腎障害などを引き起こす。レプトスピラは環境中での濃度が低くて検出が難しく、また、環境サンプルから単離・培養をすることも比較的困難。そのため、レプトスピラが野外でどのように分布し生息しているかについては、多くが未解明であり、レプトスピラ症の感染リスクを評価することも容易ではなかった。
水中の「環境DNA」を分析、川の水から直接検出に成功
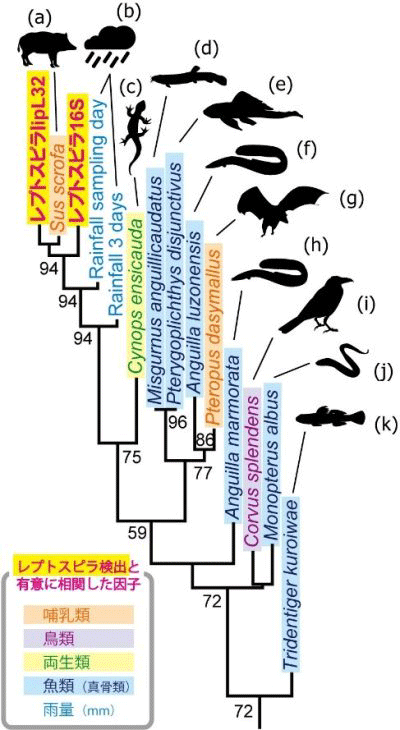
今回、研究グループは、水中に存在する「環境DNA」に着目し、河川水から直接レプトスピラのDNAを検出することに成功した。夏季(7月~10月)にレプトスピラ症の発生が報告されている沖縄島の河川で採水を行い、ろ過により懸濁物を高度に濃縮してから、DNAを抽出。抽出された環境DNAから、レプトスピラが保有する遺伝子(lipL32と16S rRNA)をPCRで増幅し、そのDNA配列を次世代シークエンサーで大量解析した。
その結果、病原性のLeptospira alstoniiや中間型のL. wolffiiなど6種のレプトスピラが検出され、そのDNA配列数が採水時の雨の量と有意に相関することも確認された。さらに、同じ環境DNAサンプルを用いて、レプトスピラと同時に出現する動物のDNAを分析したところ、レプトスピラの保菌動物であることが知られるイノシシやオオコウモリなど10種の動物を特定することにも成功した。また同研究では、同じ手法で細菌類全般を対象としたDNA分析も行っており、河川水中でレプトスピラと同時に出現する12種の環境細菌を検出。その中には、実験下でレプトスピラと共にバイオフィルムを形成することが指摘されているSphingomonas類が含まれた。保菌動物から環境中へ排出されたレプトスピラは、他の共生細菌と共に凝集して環境中に存続し、雨や濁流などによって再び河川水中に拡がるということが推察されるという。
以上の研究結果から、レプトスピラ症の予防においては、雨による濁流が見られる川や、イノシシなどの野生哺乳類が生息する付近の水場や泥土を避けるのが望ましいということが示唆された。また、この研究によって開発された、環境DNA分析に基づくレプトスピラの直接検出と保菌動物候補の推定方法は、沖縄県だけでなくさまざまな国や地域にも適用できる。そのため同研究の成果は、レプトスピラ症の環境リスク評価、保菌動物の管理、衛生環境の改善などに役立てられることが期待されると、研究グループは述べている。
▼関連リンク
・琉球大学 研究成果



