淋菌感染症治療に用いられるスペクチノマイシン
産業技術総合研究所と北海道大学は4月4日、環境バクテリアの16SリボソーマルRNA(16S rRNA)遺伝子ライブラリーの中から、淋菌感染症治療に用いられる抗生物質のスペクチノマイシンに対して耐性を示す16 SrRNA遺伝子を複数発見し、新たな耐性変異を発見したと発表した。この研究は、産総研生物プロセス研究部門合成生物工学研究グループの宮崎健太郎研究グループ長と、北大大学院理学研究院の北原圭特任助教らの研究グループによるもの。研究成果は英科学雑誌「Scientific Reports」に掲載されている。
rRNAは、抗生物質の主要なターゲットのひとつ。rRNAに耐性変異が生じると、抗生物質が効かなくなる。そのため、耐性変異の情報は耐性菌の早期発見に有用だが、これまでrRNAの耐性変異の同定は難しく、耐性変異に関する情報は十分に蓄積されていなかった。
今回研究グループは、産総研が開発した16S rRNAを生物種間で交換する手法を用いて、環境中のさまざまなバクテリア由来の16S rRNAの中から、16S rRNAに作用する抗生物質のひとつであるスペクチノマイシンに対して耐性を示す16S rRNA遺伝子をスクリーニング。発見した耐性16S rRNA遺伝子の変異解析による新たな耐性変異の同定に取り組んだ。
スペクチノマイシンに対する新たな3つの耐性変異も発見
研究グループは、まず、大腸菌16S rRNA遺伝子欠損株が生育できる環境バクテリア由来の16S rRNA遺伝子を大量に取得し、数万種の大腸菌変異株からなるライブラリーを作製。次に、この大腸菌変異株ライブラリーを、スペクチノマイシンを含む培地で培養し、生育できた大腸菌変異種すなわち耐性を示す大腸菌変異種を分離した。これらの16S rRNA遺伝子の解析により、4種のスペクチノマイシン耐性16S rRNA遺伝子を同定したという。それらの遺伝子の変異解析により、新たにスペクチノマイシンに対する3つの耐性変異を発見したとしている。
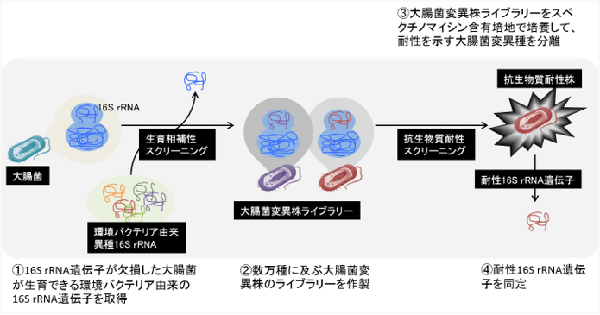
画像はリリースより
これまで、バクテリアの複数のrRNAを個別に調べて抗生物質耐性を持つものを特定できる有効な手段はなかったが、今回用いた産総研独自の手法であれば個々の16S rRNAの機能を調べて抗生物質耐性を持つものを特定できることがわかった。この手法はスペクチノマイシン以外の抗生物質にも適用できるため、この手法でさまざまな抗生物質の耐性スクリーニングを行い耐性変異に関するデータを蓄積すれば、耐性菌の早期発見・診断に有用な抗生物質耐性変異のデータベースの構築も可能、と研究グループは述べている。
▼関連リンク
・産業技術総合研究所 研究成果



