1コピーあたりから発現するタンパク質量が減少する遺伝子を同定する実験法を開発
岡山大学は2月16日、同大学異分野融合先端研究コアの守屋央朗准教授と同大学大学院自然科学研究科博士後期課程の石川浩史大学院生らの研究グループが、酵母細胞を用いた研究によって、遺伝子発現の乱れをタンパク質分解により調整するメカニズムを明らかにしたと発表した。この研究は、カリフォルニア大学バークレー校のNicholas Ingolia助教と岩崎信太郎研究員(現・理化学研究所准主任研究員)との共同研究により行われ、米国オンライン科学誌「PLOS Genetics」に掲載された。
画像はリリースより
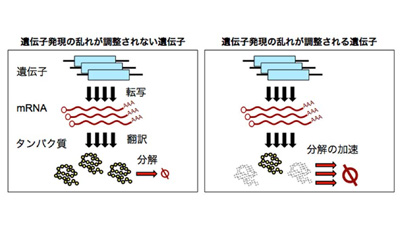
通常、遺伝子コピー数の増加は、そのままタンパク質量の増加に結び付く。一方、コピー数が増加してもタンパク質量の増加に反映されない遺伝子が知られており、それらの発現プロセスには何らかの調整メカニズムがあると考えられる。
研究グループは、このような調整を受ける遺伝子を体系的に探索するため、コピー数を人工的に増やしたときに1コピーあたりから発現するタンパク質量が減少する遺伝子を同定する実験法を開発し、第一染色体の54の遺伝子を調べた。その結果、5つの遺伝子を同定し、それらすべてが複合体を構成するタンパク質(サブユニット)をコードしていることを見出した。さらに、それらの複合体に含まれる他のサブユニットを解析したところ、そのほとんどが調整を受けたことから、この調整は複合体を作るタンパク質をコードする遺伝子に対して行われていることがわかった。さらにこれらの結果から、酵母が持つ遺伝子のうち約10%が調整を受けると見積もれたとしている。
サブユニットの量比の乱れ、どのように感知され分解に導かれるのかの解明へ
次に、この調整が、発現プロセスのどこで起きているかを調べた。その結果、調整を受けるタンパク質の遺伝子コピー数を増加させたとき、mRNAの量とタンパク質の合成速度は変化しない一方で、タンパク質の分解速度が増加していることがわかった。タンパク質の合成速度の解析は、共同研究者のIngolia博士らによって近年開発された「リボソームプロファイリング」と呼ばれる革新的な技術を用いることで実現。さらに、この分解速度の増加が、細胞内の主要なタンパク質分解経路、「ユビキチン−プロテアソーム系」によるものであることも明らかになった。
細胞内には多数の複合体が存在している。複合体を作るサブユニットの量比が揃わなければ、複合体が異常な構造や機能を持ち、細胞全体の機能に悪影響を及ぼすことが知られている。この研究で見出されたことは、遺伝子発現の乱れにより生じるサブユニットの量比のアンバランスを解消し、細胞の機能を正常に保つためのメカニズムだと考えられる。
今回の研究は、遺伝子発現の乱れが細胞全体に影響を及ぼすことを防ぐ頑健性を生命システムがどのように達成するかという、生命の基本的な理解を深めるもの。サブユニットの量比の乱れがどのように感知され分解に導かれるのかを今後明らかにしていきたいと、研究グループは述べている。
▼関連リンク
・岡山大学 プレスリリース






