「3層オミックス参照パネル」として公開
日本医療研究開発機構(AMED)は4月14日、岩手医科大学いわて東北メディカル・メガバンク機構の生体情報解析部門を中心とした研究チームが、エピゲノム(遺伝子修飾情報)の1つであるDNAメチル化に着目し、2種類の血液細胞(単球とCD4陽性Tリンパ球)における100人規模の全DNAメチル化情報の高精度な解析に初めて成功したと発表した。
画像はリリースより
この研究は、東北大学東北メディカル・メガバンク機構との共同で行われたもの。今回解析したエピゲノム(DNAメチル化情報)は、ゲノム(遺伝子情報)・トランスクリプトーム(遺伝子転写情報)と合わせ、3層のオミックス(網羅的生体分子情報)参照パネルとして公開された。
近年、ゲノムの塩基配列を変えずにその周辺(エピ)を修飾することで発現を制御する「エピゲノム」が注目されている。エピゲノムの1つであるDNAメチル化は、細胞分化の仕組みの主役である一方、喫煙・飲酒・ストレスなどのさまざまな環境要因によって異常が生じ、生活習慣病・がん・うつなどの種々の疾患を引き起こす誘因であることが明らかになっている。しかし、日本人のDNAメチル化の状態や個人、細胞ごとの違いを大規模・網羅的に調査した研究は、これまでになかったという。
2400万箇所のDNAメチル化部位を同定、個人ごと、細胞ごとの違いを明らかに
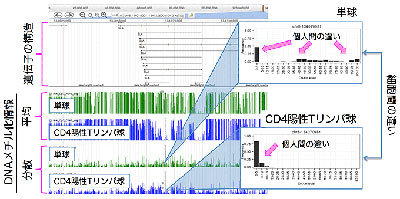
そこで、同研究グループは、約100名の人における全DNAのメチル化を詳細に解析。エピゲノム解析の対象となる組織には皮膚、口腔粘膜、血液などがあるが、汎用性が高く多くの疾患との関連性が指摘されている血液細胞を用いたという。
血液から細胞をレーザーで判別する「セルソーター」を用いて血液細胞を一つひとつ回収し、98%以上という高い純度で種々の血液細胞を分別収集。その中から、エピゲノムの特徴が大きく異なる「単球」と「CD4陽性Tリンパ球」という2つの血液細胞から、DNAとRNAを取り出し、次世代シークエンサーを用いて、DNAの塩基配列の情報、遺伝子転写の情報(トランスクリプトーム)に加え、DNAメチル化の情報(を解析した。その結果、2400万箇所のDNAメチル化部位を同定するとともに、個人ごとの違いや細胞ごとの違いを明らかにした。
この研究の成果により、さまざまな環境要因や疾患におけるエピゲノム異常の同定が可能となることが期待されるという。同研究グループは、今後も追跡調査を行い、生活習慣の変化や疾患の発症とエピゲノムとの関連性を明らかにすることで、個別化医療・個別化予防の実現につなげていきたいと述べている。
▼関連リンク
・日本医療研究開発機構 プレスリリース






