ES細胞とiPS細胞で発現する全RNAの網羅的な解析を実施
理化学研究所は3月11日、ES細胞とiPS細胞で発現する全てのRNAを比較解析し、ES細胞で発現しているノンコーディングRNA(ncRNA)の多くが、iPS細胞では十分に発現していないことを発見したと発表した。
画像はプレスリリースより
この研究は、理研ライフサイエンス技術基盤研究センター トランスクリプトーム研究チームのピエロ・カルニンチ チームリーダー、アレクサンダー・フォート客員研究員と、理研統合生命医科学研究センター免疫器官形成研究グループの古関明彦グループディレクターらの研究グループによるもの。日本学術振興会「最先端・次世代研究開発支援プログラム」などの支援を受けて行われ、成果は米科学雑誌「Cell Cycle」オンライン版に2月12日付けで掲載されている。
これまで、ES細胞とiPS細胞では、幹細胞としての性質の多くが共通している一方で、遺伝子発現の解析からは異なる状態にある可能性が報告されていた。ただし、これらの解析は主にタンパク質の設計図となるメッセンジャーRNA(mRNA)や、既知のノンコーディングRNA(ncRNA)だけを解析したもので、ES細胞とiPS細胞のncRNAを網羅的に解析する研究は行われていなかった。
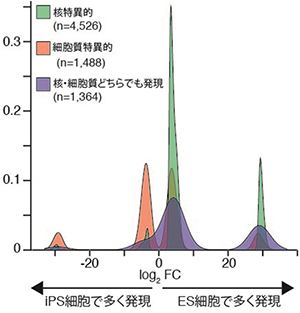
レトロトランスポゾン由来のRNA配列などが十分に発現せず
同研究グループは2014年、転写産物(RNA)の網羅的な解析を定量的かつ高感度に行うことができる理研の独自技術「CAGE法」を用い、iPS細胞とES細胞の核内を研究。すると、これまで知られていなかった数千種類のRNA(幹細胞特異的な転写産物:NASTs)が発現していることが見いだされた。そして、その多くがncRNAであることが明らかになったという。今回の研究でも同様の解析手法を用い、ES細胞とiPS細胞で異なる発現を示すRNAの解析を試みた。
実験では、マウス由来のES細胞とiPS細胞を用い、ncRNAを含めた全転写産物の網羅的な発現比較を行った。その結果、ES細胞の核内で発現するncRNAの多くが、iPS細胞では十分に発現していないことが分かったという。これらのncRNAの中には、多能性の維持に関わる遺伝子の発現を促進する遺伝子制御部位や、多能性に関わるレトロトランスポゾン由来のRNA配列が含まれていた。これは、既存のiPS細胞作製法では、ES細胞で機能している多くの遺伝子制御部位の活性化が十分に起きていないことを示唆しているという。
理研は今後、iPS細胞作製技術の改良や、臨床応用に適したiPS細胞の評価選別法の確立に役立つと期待を寄せている。
▼外部リンク
・理化学研究所 プレスリリース




