膨大な数の遺伝子からカギとなる遺伝子の発現量変化を抽出
理化学研究所は12月17日、生命システム研究センター 多階層生命動態研究チームの古澤力チームリーダー、鈴木真吾研究員らの研究チームが、複数の抗生物質に対して耐性を持つ大腸菌の解析を行い、少数遺伝子の発現量データだけで、抗生物質への耐性を定量的に予測できる新手法を開発したと発表した。
画像はプレスリリースより
この手法によって、バクテリアが抗生物質への耐性を獲得する際、カギとなるのはどの遺伝子の発現量変化なのか、膨大な数の遺伝子から抽出することが可能になるという。同研究の成果は、英国の科学雑誌『Nature Communications』オンライン版に12月17日付で掲載されている。
耐性獲得を抑制する手法、新規抗生物質への開発貢献に期待
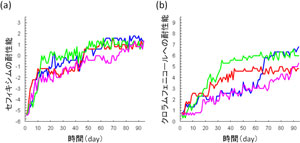
研究チームは、さまざまな抗生物質を添加した環境で、大腸菌を長期に植え継いで培養。生体内で行われる抗生物質に対する耐性獲得の進化プロセスを、生体外で再現できる実験システムを構築した。
実験では、どの遺伝子に突然変異が起きるのか、発現量に変化が生じるのかを調べると同時に、各種薬剤に対する耐性の変化を詳細に解析。その結果、ひとつの抗生物質への耐性獲得が他の抗生物質に対する耐性獲得を引き起こす一方、別の抗生物質に対しては、耐性の低下を引き起こすことが明らかになったという。
さらに全く種類の異なる遺伝子の変異が、類似の機能を持つ遺伝子の発現量の変化を引き起こし、それが抗生物質耐性の獲得につながっていることが示唆されたとしている。
これらの手法により、どの遺伝子がどの抗生物質への耐性獲得に寄与しているかを定量的に解析することが可能となり、耐性獲得を抑制する手法の開発や新規抗生物質の開発への貢献が期待できる。(大場真代)
▼外部リンク
・理化学研究所 プレスリリース




