がん抑制機能を持つほかは、詳細な機能が不明だったmicroRNA-34a
東京医科歯科大学は3月23日、がんなど様々な疾患において重要な働きを担うmicroRNA(miRNA)の標的遺伝子を同定する新たな方法を開発し、がん抑制miRNA-34aの新たな標的遺伝子として、乳がんの増悪化に関わるGFRA3を同定したと発表した。この研究は、同大学大学院医歯学総合研究科システム発生・再生医学分野の浅原弘嗣教授、伊藤義晃プロジェクト助教の研究グループが、国立成育医療研究センター、名古屋市立大学、インペリアル・カレッジ・ ロンドン、カリフォルニア工科大学およびベックマン研究所と共同で行ったもの。同研究成果は、国際科学誌「Proceedings of the National Academy of Sciences USA」(米国科学アカデミー紀要)に掲載されている。
画像はリリースより
miRNAの機能を明らかにする上で、その標的遺伝子を同定することは最も重要だ。しかし、従来の標的遺伝子探索方法には不十分な点がいくつかあった。マイクロアレイやRNA-sequenceなどのmRNAの発現変化を網羅的に解析するトランスクリプトーム解析と、miRNAの配列情報からターゲットを予測するTargetScanなどの予測ツールを用いた解析を組み合わせてスクリーニングする方法が一般的だが、 miRNAはmRNAの不安定化だけでなく、翻訳抑制を介して標的遺伝子を抑制するため、タンパク質レベルではなくmRNAレベルでの発現変化を観察するトランスクリプトーム解析では、翻訳抑制される標的遺伝子を同定するには十分ではない。また、TargetScanなどの予測ツールは、ターゲットとなるmRNAの3’UTRのみの情報で予測する場合が多く、近年報告されている、コーディング領域や5’UTRを介したmiRNAの制御について、標的遺伝子を同定することができないという。
microRNAの標的遺伝子の新たな同定法を開発
今回の研究では、5’UTR、コーディング領域、3’UTRを含む全長遺伝子配列をレポーターとして用いて、タンパク質レベルでの発現変化を測定できるルシフェラーゼアッセイをベースにした、短期間で効率的にmiRNAの標的遺伝子を同定するレポーターライブラリーシステムと呼ばれる手法を開発。同手法を用いて、がんを抑制する機能をもつmiRNAであるmiR-34aについて、標的遺伝子の探索を行ったという。
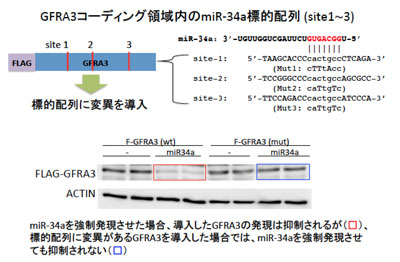
その結果、既に報告されている標的遺伝子に加え、新規な標的遺伝子としてGFRA3、FAM76A、REM2およびCARKLを同定。また、miRNAは主に標的遺伝子の3’UTRを介して制御するとされているが、GFRA3はタンパク質をコードしているコーディング領域(CDS)を介して直接制御されていることが明らかになったという。
このGFRA3を、MDA-MB-231という乳がん細胞においてノックダウンすると、MDA-MB-231細胞の増殖が抑制された。また、GFRA3の発現が高い乳がんを持つ患者は、GFRA3の発現が低い乳がんを持つ患者に比べて生存率が低いことがわかったという。これらの結果から、GFRA3は乳がん増悪化に関わる重要な遺伝子であることが明らかになった。
miRNAはがんだけでなく、様々な疾患に関与していることが明らかになりつつあるが、同手法を用いたmiRNAの標的遺伝子の同定により、がんやその他の疾患の病態解明に寄与することが期待されると、研究グループは述べている。
▼関連リンク
・東京医科歯科大学 プレスリリース






