約10万件のバイオビッグデータを有効活用する方法を研究
九州大学は11月9日、世界中から報告されたタンパク質とゲノムDNAの結合情報を全てデータベース化し、組織や臓器を形成する司令塔的なタンパク質の探索に応用できることを示したと発表した。この研究は、同大大学院医学研究院の沖真弥助教と目野主税教授が、情報システム研究機構・ライフサイエンス統合データベースセンター(DBCLS)の大田達郎特任研究員などと共同で行ったもの。研究成果は、国際学術雑誌「EMBO Reports」オンライン版に掲載されている。
画像はリリースより
ヒトの細胞の中に存在するゲノムDNAは、ヒストンと呼ばれるタンパク質に巻きついてクロマチンという構造を形成している。転写因子と呼ばれる約1,000種類のタンパク質がクロマチンに結合することで、付近の遺伝子発現のオン・オフを制御し、組織や臓器を形成し、正常に機能させている。さらに、これらに異常があると遺伝子の制御が乱れ、胎児の奇形、がんや自己免疫疾患などの原因になると言われている。しかし、転写因子が30億塩基対もあるゲノムDNAのどこに結合しているのかということについては明らかにされていなかった。その後、2007年にChIP-seq法という手法が開発され、これまで10万件近くもの実験結果が報告されているが、データ量が膨大で、ビッグデータから必要な知識を取り出すことは困難だった。
免疫細胞などを形成する司令塔的な転写因子の予測に
研究グループは、国立遺伝学研究所のスーパーコンピューターシステムを利用して約10万件のChIP-seqビッグデータを全て収集・計算し、ヒストンや転写因子がゲノムDNAに結合する位置情報をすべて可視化することに成功。また、実験に用いられた試料に関する情報を整理し、全てのデータに実験対象の細胞や転写因子の名前を明記した。これらのデータは2015年12月よりChIP-Atlas(https://chip-atlas.org)というWebサービスとして公開したが、その後、更新や改訂を重ね、今回正式に論文として報告した。
ChIP-seqデータは、高校で習う程度の転写因子やゲノムに関する基礎知識があれば誰でも使うことが可能。地図帳をめくる感覚で遺伝子にアクセスし、そこに結合する転写因子やヒストンの種類、および細胞タイプなどを視覚的に理解できるという。ChIP-seqのビッグデータは、世界中から毎月約1,500件も報告されているが、これらのデータは毎月ChIP-Atlasに追加されているため、利用者は常に最新のデータを閲覧することが可能。毎月、国内外から約10万PVのアクセスがあり、これまで約50報の論文に引用されている。主に遺伝子発現を制御するしくみに関する研究のほか、薬の作用、がん、老化、生物進化などにかかわる転写因子の研究に応用。これらの利用事例は今回の論文にも述べられており、今後も幅広い生命科学研究の発展に寄与できると思われるとしている。
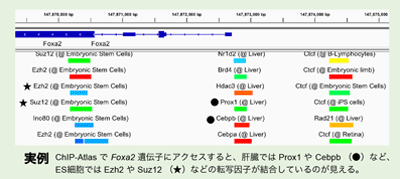
続けて研究グループは同データを使用し、組織特異的に発現する複数の遺伝子が、どの転写因子によって制御されているのかについて解析。HNF4AやFOXA2などの転写因子が肝臓特異的遺伝子の多くに結合し、これらが肝臓で重要な複数の遺伝子の発現をまとめて制御する司令塔的役割を果たしていることを明らかにした。さらに、血管内皮細胞特異的遺伝子にはTAL1, FOS, JUNなど、マクロファージ特異的遺伝子群にはSTAT1やSPI1などの転写因子が高頻度に結合し、それぞれの細胞タイプの司令塔的な転写因子であることも示唆。同データが免疫細胞、肝臓、血管などを形成する司令塔的な転写因子の予測に応用可能であることを示した。これらの成果は、それぞれの細胞タイプの司令塔的な転写因子であると考えられ、将来的にはダイレクト・リプログラミング技術による組織再生への応用が期待されるという。
▼関連リンク
・九州大学 プレスリリース




